Conformément aux conditions d’utilisation de la plateforme Infoscience, les métadonnées d’Infoscience sont placées sous licence Creative Commons CC0 1.0. Cette licence permet l’utilisation, la modification et la redistribution des métadonnées sans nécessiter de consentement préalable. Le modèle de données d’Infoscience est décrit à cette adresse.
En tant qu’utilisateur.trice final.e, vous êtes autorisé.e à utiliser les œuvres mises à disposition sur Infoscience, à condition de respecter les droits d’auteur.trice et les termes de la licence associée.
Cela inclut :
- Citer correctement les auteur.trices,
- Fournir des mentions bibliographiques complètes (titre, éditeur, année de publication, lien permanent : Handle),
- Appliquer les conditions d’une licence copyleft si spécifiée, telle que Creative Commons.
Toute utilisation des contenus d’Infoscience à des fins autres que personnelles et non commerciales nécessite l’autorisation expresse des détenteur.trices des droits. Les œuvres restreintes à la communauté EPFL ne peuvent être utilisées, téléchargées ou distribuées en dehors de ce cadre sans une autorisation formelle des auteur.trices.
Il vous incombe, en tant qu’utilisateur.trice final.e, d’évaluer de manière appropriée les droits d’auteur.trice et les aspects juridiques liés à l’utilisation des contenus Infoscience.
Vous êtes responsable de l’usage que vous faites de ces informations et devez assumer pleinement cette responsabilité.
■ Exporter les métadonnées
Infoscience propose divers formats d’export de références bibliographiques afin de permettre aux utilisateur.trices de transférer facilement ces données vers différents systèmes ou services, comme des logiciels de gestion bibliographique tels que Endnote ou Zotero. Ces formats respectent les standards de métadonnées largement utilisés tels que DataCite, BibTeX, et RIS.
De plus, Infoscience propose également d’autres formats d’export structurés tels que CSV, JSON, et XML, offrant ainsi une flexibilité accrue pour répondre aux besoins spécifiques des utilisateur.trices et des systèmes.
Il est important de souligner que ces services d’export sont accessibles sans nécessiter d’authentification préalable.
En allant sur une liste de résultat, cliquez sur le bouton “Exporter” (1)
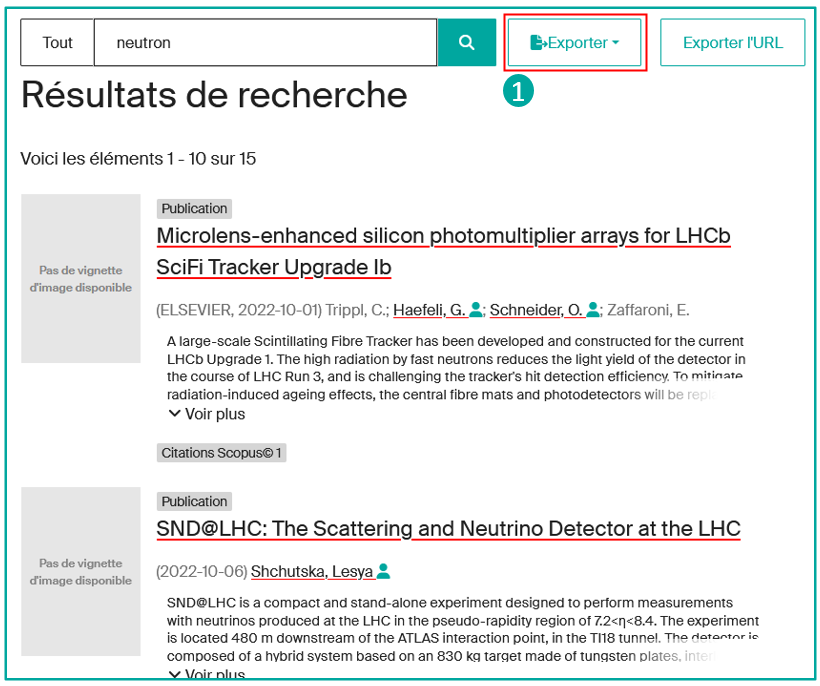
Actuellement, Infoscience ne permet pas d’exporter l’ensemble des résultats en une seule opération. Vous devez préalablement sélectionner la catégorie de contenu que vous souhaitez exporter, par exemple, les résultats de type “Publication“, “Dataset ou Produit“, ou “Brevet“.
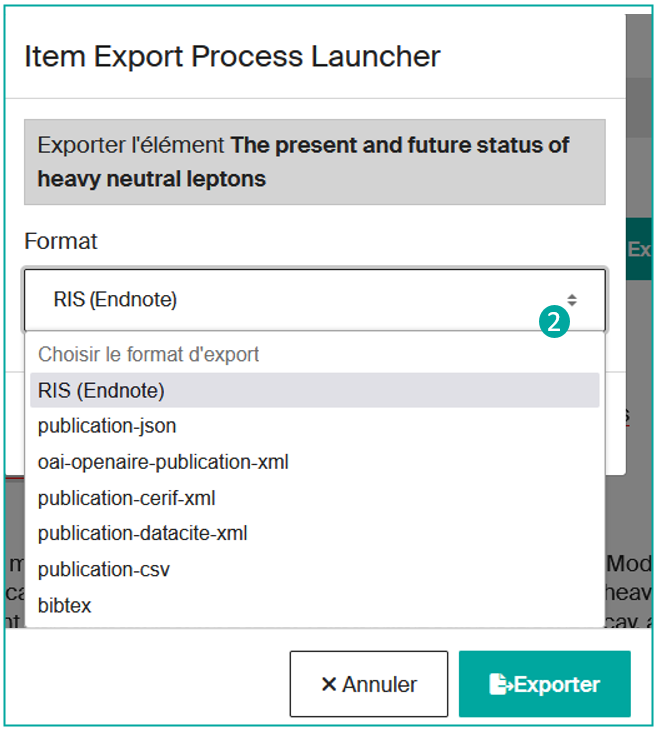
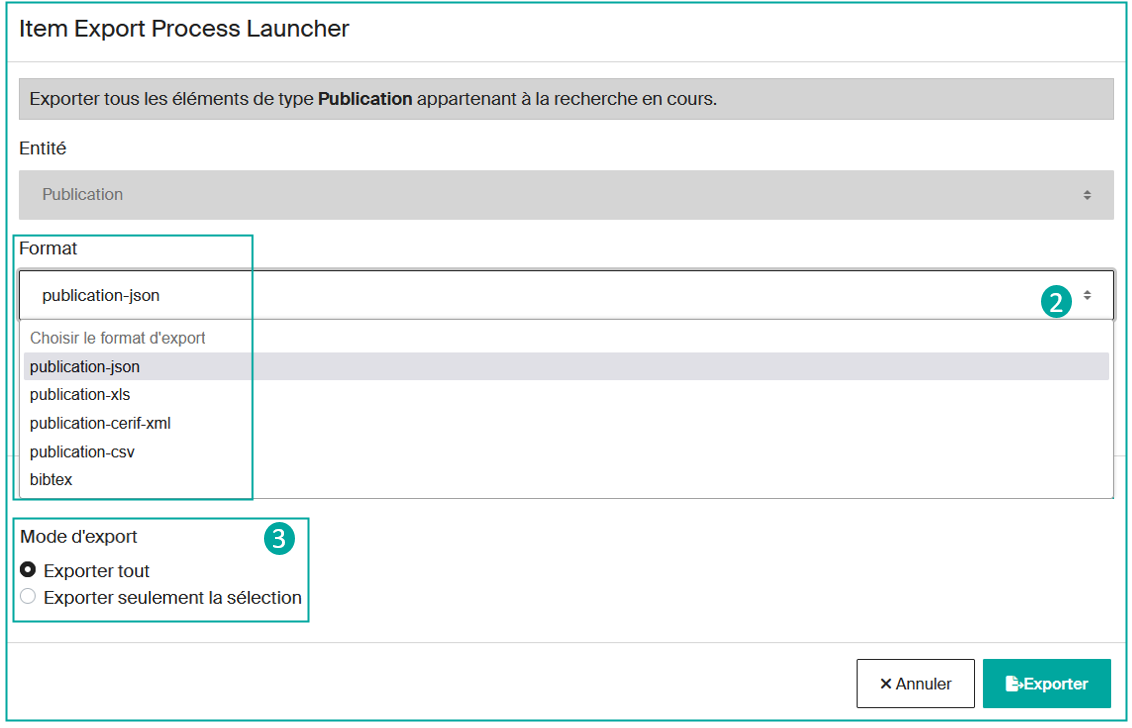
Après avoir choisi la catégorie des résultats à exporter, vous devrez déterminer le format d’export (2) souhaité parmi les options disponibles (json, xls, csv, bibtex, etc.).
Ensuite, vous pouvez décider d’exporter tous les résultats ou seulement d’exporter une sélection manuelle (3).
Il est important de noter que l’exportation de tous les résultats peut prendre un certain temps, en fonction du volume de données à traiter.
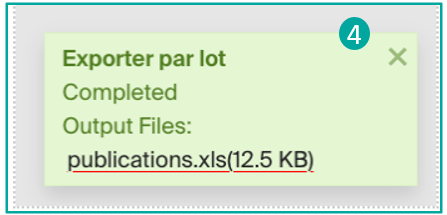
Une fois votre choix effectué, cliquez sur le bouton “Exporter” pour démarrer le processus. Une fois ce dernier terminé, vous pourrez récupérer le fichier généré (4).
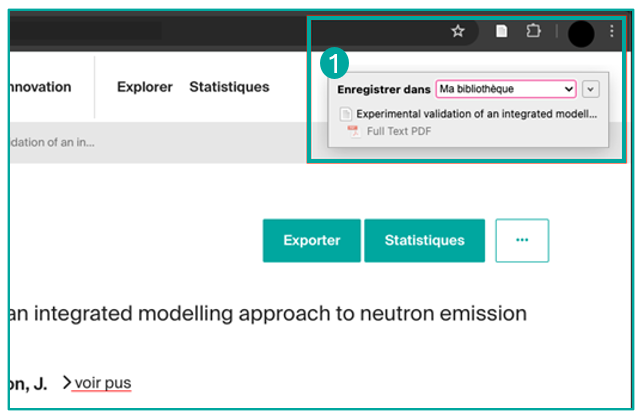
Vous pouvez aisément enregistrer des références bibliographiques depuis une notice infoscience.
Pour ce faire, utilisez l’extension Zotero connector et cliquez sur le bouton “Save to Zotero” de votre navigateur, afin de capturer et de sauvegarder la référence directement dans votre bibliothèque Zotero (1).
■ Réutiliser les métadonnées
L’API REST
Endpoint : https://infoscience.epfl.ch/server/api
Infoscience est une solution entièrement basée sur des services back-end fournis par une API de type REST. Cette API repose sur plusieurs standards pour assurer une interaction fluide et auto-documentée : ALPS (Application Level Profile Semantics), HATEOAS (Hypertext As The Engine Of Application State) et HAL (Hypertext Application Language). Un navigateur dédié HAL Browser fournit une description complète de tous les endpoints disponibles.
Les réponses de l’API sont fournies dans un format standard exprimé en JSON. De plus chaque réponse de l’API inclut des liens vers les opérations suivantes disponibles, permettant à l’API de se décrire elle-même et de guider l’utilisateur.trice dans ses interactions.
L’API permet un accès en lecture seule aux métadonnées et fichiers publics sans nécessiter d’authentification.
Cela permet à n’importe quel utilisateur.trice de consulter les informations disponibles publiquement.
Les utilisateur.trices peuvent obtenir un token activable depuis leur compte sur la plateforme Infoscience, qui leur accorde des droits d’accès spécifiques basés sur le ou les rôles qui leurs sont attribués.
Ce token est essentiel pour effectuer des requêtes authentifiées à l’API et pour accéder aux ressources protégées.
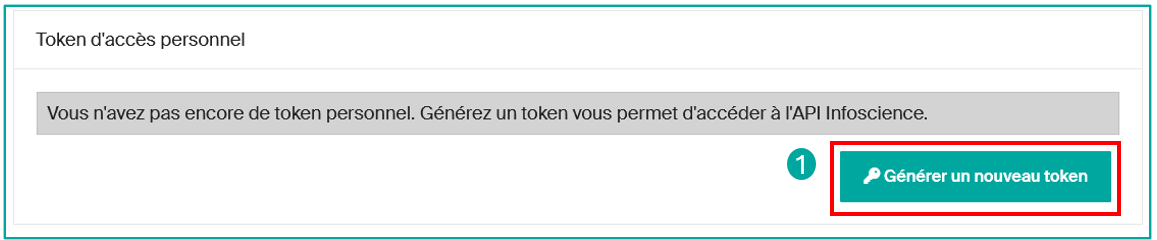
- Connectez-vous à votre compte sur la plateforme Infoscience.
- Cliquez sur Compte et profil sur l’icône de votre profil
- Sur la page de votre profil, cliquez sur “Générer un nouveau token” (1).
- Notez et conservez le token affiché dans un endroit sécurisé.
Important : Une fois généré, le token ne sera plus visible. Si vous le perdez, vous devrez en générer un nouveau.
Il est possible de créer un “compte service” (local) non rattaché à un compte individuel pour bénéficier de droits supplémentaires. Dans des contextes spécifiques et justifiés, ces comptes peuvent également obtenir des droits en écriture.
Pour toute demande, merci de contacter l’équipe Infoscience.
Pour garantir la sécurité de votre token, veuillez suivre les recommandations suivantes :
- Ne partagez jamais votre token : Ne transmettez jamais votre token à un tiers, car cela pourrait compromettre la sécurité de vos accès.
- Évitez l’exposition : Ne l’incluez pas dans du code source partagé ou des dépôts publics (par exemple, sur GitHub).
- Si vous suspectez que votre token a été compromis ou si vous l’avez perdu, vous pouvez en générer un nouveau en suivant les mêmes étapes que pour la première obtention.
Vous trouverez la documentation officielle de l’API basée sur la solution DSpace et la distribution DSpace-CRIS à ces adresses :
- DSpace : https://github.com/DSpace/RestContract
- Endpoints spécifiques à la distribution DSpace-CRIS : https://github.com/4Science/Rest7Contract
Vous trouverez ci-dessous quelques exemples de requêtes réalisées avec l’API. Afin de connaitre les index de recherche disponibles, n’hésitez pas à vous référer au modèle de données disponible à cette adresse.
Get item:
curl –location ‘https://infoscience.epfl.ch/server/api/core/items/{{uuid_item}}’ \
–header ‘accept: application/json, text/plain, */*’ \
–header ‘Authorization: Bearer VOTRE_TOKEN’
Get item with bundles/bitstreams:
curl –location ‘https://infoscience.epfl.ch/server/api/core/items/{{uuid_item}}?embed=bundles/bitstreams‘ \
–header ‘accept: application/json, text/plain, */*’ \
–header ‘Authorization: Bearer VOTRE_TOKEN’
Search items with ‘artificial intelligence’ criteria:
curl –location ‘https://infoscience.epfl.ch/server/api/discover/search/objects?sort=score,DESC&page=0&size=20&configuration=researchoutputs&query=Artificial+intelligence‘ \
–header ‘accept: application/json, text/plain, */*’ \
–header ‘Authorization: Bearer VOTRE_TOKEN’
Search items affiliated with a specific unit:
curl –location ‘https://infoscience.epfl.ch/server/api/discover/search/objects?sort=dc.date.issued,DESC&page=0&size=10&configuration=researchoutputs&query=unitOrLab:LASUR‘ \
–header ‘accept: application/json, text/plain, */*’ \
–header ‘Authorization: Bearer VOTRE_TOKEN’
L’entrepôt OAI
Infoscience est compatible avec le protocole OAI-PMH (Open Archives Initiative Protocol for Metadata Harvesting), un standard qui permet l’échange automatisé de métadonnées entre entrepôts de données. Il facilite l’accès et la gestion des informations sur les ressources numériques.
L’entrepôt OAI-PMH d’Infoscience est accessible à l’adresse suivante : https://infoscience.epfl.ch/server/oai/openaire4
L’entrepôt Infoscience supporte plusieurs formats de métadonnées notamment :
- Dublin Core (oai_dc)
- MarcXML (marcxml)
- OpenAIRE Guidelines for Literature, Institutional, and Thematic Repositories (oai_openaire)
| Set Spec | Set Name | Description |
| OpenAIREv4 | OpenAIREv4 | Documents exposés vers OpenAIRE |
| fulltext-public | fulltext-public | Documents en open access |
| fulltext | fulltext | Documents avec fulltext |
| theses-bn | theses-bn | Thèses de doctorats soutenues à l’EPFL |
| doi | DOI | Documents avec un DOI attribué par l’EPFL |
| col_20.500.14299_2 | Books and Book parts | Tous les documents de la collection Books and Book parts |
| col_20.500.14299_3 | Conferences, Workshops, Symposiums, and Seminars | Tous les documents de la collection Conferences, Workshops, Symposiums, and Seminars |
| col_20.500.14299_16 | Contents | Tous les documents de la collection Contents |
| col_20.500.14299_4 | Datasets and Code | Tous les documents de la collection Datasets and Code |
| col_20.500.14299_5 | EPFL thesis | Tous les documents de la collection EPFL thesis |
| col_20.500.14299_6 | Images, Videos, Interactive resources, and Design | Tous les documents de la collection Images, Videos, Interactive resources, and Design |
| col_20.500.14299_7 | Journal articles | Tous les documents de la collection Journal articles |
|
col_20.500.14299_8 |
Newspaper, Magazine, or Blog post | Tous les documents de la collection Newspaper, Magazine, or Blog post |
| col_20.500.14299_10 | Patents |
Tous les documents de la collection Patents |
| col_20.500.14299_11 | Preprints and Working Papers | Tous les documents de la collection Preprints and Working Papers |
| col_20.500.14299_12 | Reports, Documentation, and Standards | Tous les documents de la collection Reports, Documentation, and Standards |
| col_20.500.14299_13 | Student works | Tous les documents de la collection Student works |
| col_20.500.14299_14 | Teaching Materials | Tous les documents de la collection Teaching Materials |
- Notices du set fulltext-public en dublin core:
- Notices du set OpenAIREv4 au format oai_openaire:
- Notices créés ou modifiées entre le 13 et 14 juillet 2024: